
이 가운데 국내 연구진이 동물모델 대신 환자 유래 인공 미니 장기에서 얻은 데이터를 학습하는 알고리즘을 통해 항암제 반응성 예측의 정확성을 높여 주목받고 있다. 실제 사람에서의 반응에 보다 더 근접한 데이터를학습시키겠다는 것이다.
한국연구재단은 포스텍(포항공대)김상욱 교수 연구팀이 암환자 유래 인공 미니장기의 전사체 정보를 토대로 환자의 항암제 반응성을 예측하는 인공지능 기술을 개발했다고 밝혔다.
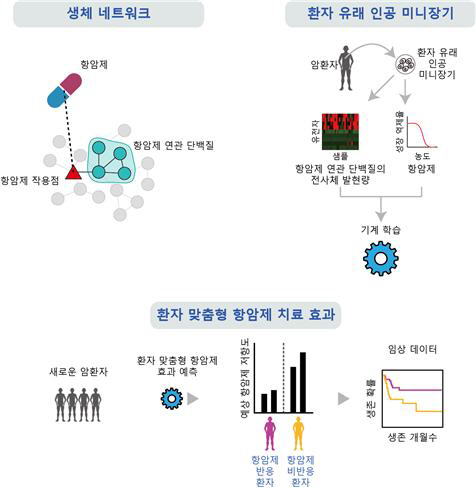
같은 암을 앓는 환자라도 항암제에 대한 반응이 다르기에 효과를 볼 수 있는 환자를 선별하는 맞춤형 치료가 중요하다.
하지만 기존 머신러닝 예측기법은 암세포의 유전체 정보를 토대로 하고 있어 정확도를 높이는 데 한계가 있었다. 불필요한 바이오마커 정보로 인해 머신러닝이 거짓 신호를 바탕으로 학습하는 문제가 있었다.
이에 연구팀은 약물의 직접적 표적이 되는 개별 단백질에 대한 전사체 정보뿐 아니라, 표적 단백질과 상호작용할 수 있는 생체 단백질 상호작용 네트워크 데이터를 이용, 예측 정확도를 높인 머신러닝 알고리즘을 소개했다.
표적 단백질로부터 기능적으로 가까운 단백질의 전사체 생성량에 대해 우선 학습하도록 한 것이다. 이를 통해 기존 머신러닝이 학습해야 했던 방대한 바이오마커 대신 선별된 바이오마커만 학습할 수 있도록 하여 정확도를 높였다.
또한 동물모델이아닌 환자 유래 미니장기의 데이터를 이용해 실제 환자에서 반응과의 차이를 좁혔다.
실제 이 방법으로 대장암에 쓰이는 5-플루오로 우라실과 방광암에 사용되는 시스플라틴 등에 대한 환자의 약물반응을 실제 임상결과와 비슷한 수준으로 예측해냈다.
항암제에 반응할 환자를 선별하는 개인 맞춤형 정밀의료 실현은 물론 새로운 항암제의 기전 규명에도 도움이 될 것으로 기대된다.
과학기술정보통신부와 한국연구재단이 추진하는 중견연구사업, 중점 연구소사업 및 포스텍 인공지능대학원 지원으로 수행된 이번 연구의 성과는 국제학술지 ‘네이처 커뮤니케이션스(NatureCommunications)’에 10월 30일 게재됐다.


